Livelli di struttura proteica
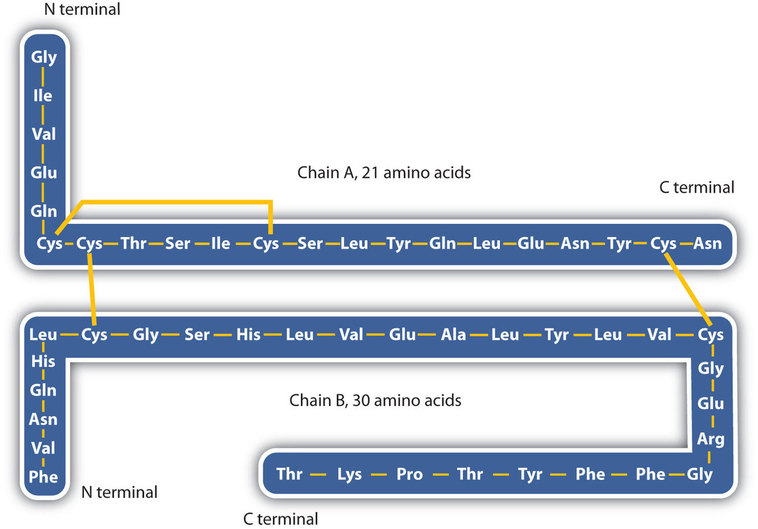
La struttura delle proteine è generalmente descritta come avente quattro livelli organizzativi. Il primo di questi è la struttura primaria, che è il numero e la sequenza di aminoacidi nella catena o nelle catene polipeptidiche di una proteina, a partire dal gruppo amminico libero e mantenuto dai legami peptidici che collegano ciascun amminoacido al successivo. La struttura primaria dell’insulina, composta da 51 aminoacidi, è mostrata in Figura \(\PageIndex{1}\).,
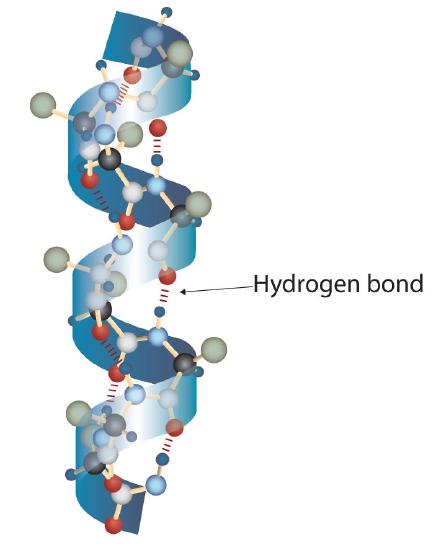
Una molecola proteica non è un groviglio casuale di catene polipeptidiche. Invece, le catene sono disposte in conformazioni uniche ma specifiche. Il termine struttura secondaria si riferisce alla disposizione fissa della spina dorsale polipeptidica., Sulla base di studi a raggi X, Linus Pauling e Robert Corey hanno postulato che alcune proteine o porzioni di proteine si torcono in una spirale o un’elica. Questa elica è stabilizzata dal legame di idrogeno intrachain tra l’atomo di ossigeno carbonilico di un amminoacido e l’atomo di idrogeno ammidico quattro amminoacidi lungo la catena (situato al turno successivo dell’elica) ed è noto come α-elica destrorsa. I dati dei raggi X indicano che questa elica fa un giro per ogni 3,6 aminoacidi e le catene laterali di questi amminoacidi proiettano verso l’esterno dalla spina dorsale arrotolata (Figura \(\PageIndex{2}\))., Le α-cheratine, presenti nei capelli e nella lana, sono esclusivamente α-elicoidali nella conformazione. Alcune proteine, come la gamma globulina, la chimotripsina e il citocromo c, hanno poca o nessuna struttura elicoidale. Altri, come l’emoglobina e la mioglobina, sono elicoidali in alcune regioni ma non in altre.
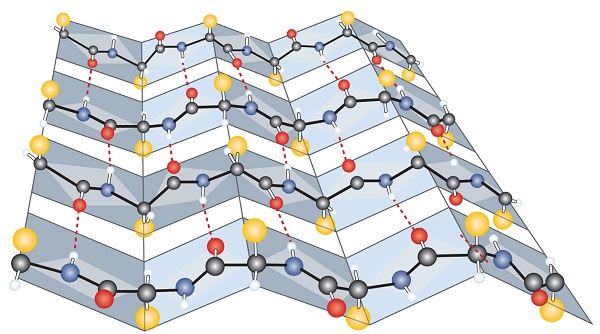
Un altro tipo comune di struttura secondaria, chiamata conformazione del foglio plissettato β, è una disposizione simile a un foglio in cui due o più catene polipeptidiche estese (o regioni separate sulla stessa catena) sono allineate l’una accanto all’altra. I segmenti allineati possono essere paralleli o antiparalleli, ovvero i terminali N possono essere rivolti nella stessa direzione su catene adiacenti o in direzioni diverse, e sono collegati da un legame di idrogeno interchain (Figura \(\PageIndex{3}\))., Il foglio β-pieghettato è particolarmente importante nelle proteine strutturali, come la fibroina di seta. È anche visto in porzioni di molti enzimi, come carbossipeptidasi A e lisozima.
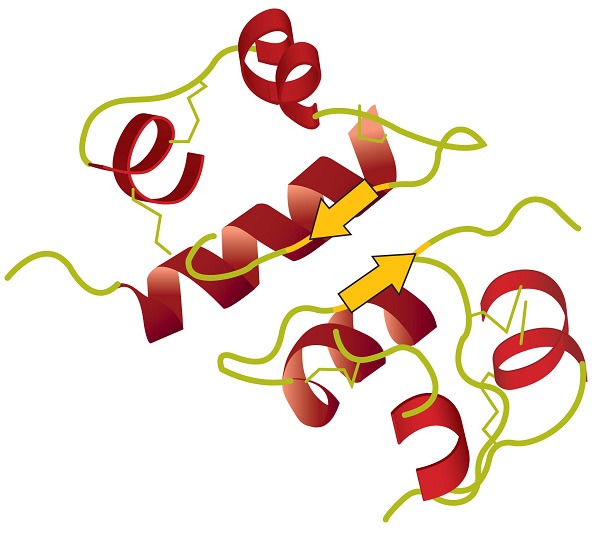
La struttura terziaria si riferisce alla forma tridimensionale unica della proteina nel suo complesso, che risulta dalla piegatura e dalla flessione della spina dorsale proteica. La struttura terziaria è intimamente legata al corretto funzionamento biochimico della proteina. La figura \(\PageIndex{4}\) mostra una rappresentazione della struttura tridimensionale dell’insulina.
Quattro tipi principali di interazioni attraenti determinano la forma e la stabilità della struttura terziaria delle proteine. Hai studiato molti di loro in precedenza.
- Legame ionico. I legami ionici derivano da attrazioni elettrostatiche tra catene laterali caricate positivamente e negativamente di aminoacidi., Ad esempio, l’attrazione reciproca tra uno aspar carbossilato acido aspartico e uno ion ammonio lisina aiuta a mantenere una particolare area piegata di una proteina (parte (a) della figura \(\PageIndex{5}\)).
- Legame idrogeno. Il legame dell’idrogeno si forma tra un atomo di ossigeno altamente elettronegativo o un atomo di azoto e un atomo di idrogeno collegato a un altro atomo di ossigeno o un atomo di azoto, come quelli che si trovano nelle catene laterali degli amminoacidi polari., Il legame idrogeno (così come le attrazioni ioniche) è estremamente importante sia nelle interazioni intra – che intermolecolari delle proteine (parte (b) della figura \(\PageIndex{5}\)).
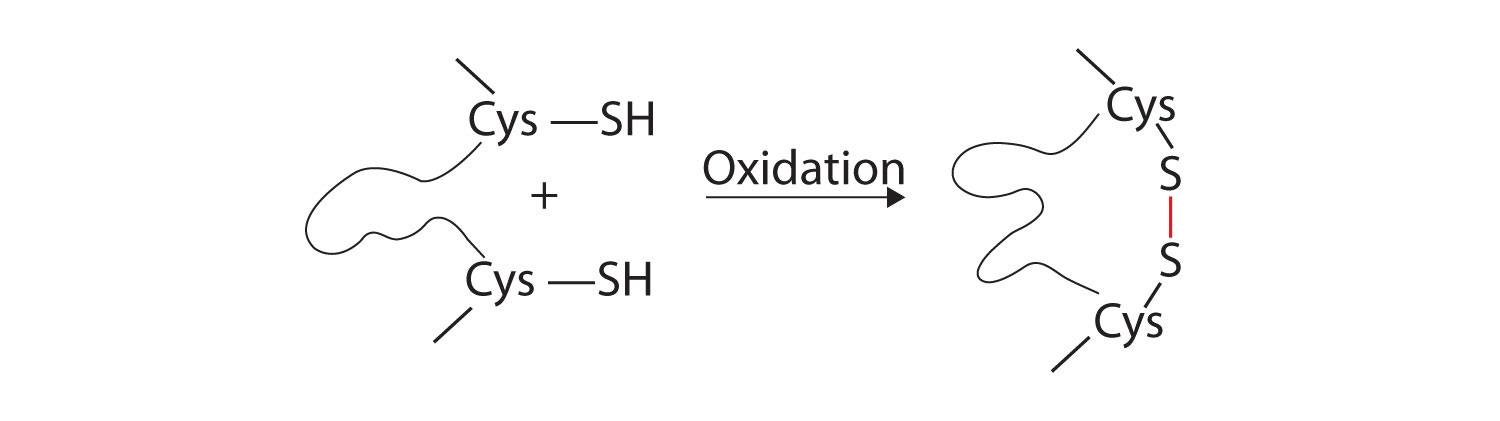
- Legami disolfuro. Due unità dell’amminoacido della cisteina possono essere portate vicino insieme come le pieghe della molecola della proteina. La successiva ossidazione e il legame degli atomi di zolfo nei gruppi sulfidrilici altamente reattivi (SH) porta alla formazione di cistina (parte (c) della figura \(\PageIndex{5}\))., Intrachain legami disolfuro si trovano in molte proteine, tra cui insulina (barre gialle in Figura \(\PageIndex{1}\)) e hanno un forte effetto stabilizzante sulla struttura terziaria.
- Forze di dispersione. Le forze di dispersione sorgono quando un atomo normalmente non polare diventa momentaneamente polare a causa di una distribuzione non uniforme di elettroni, portando ad un dipolo istantaneo che induce uno spostamento di elettroni in un atomo non polare vicino., Le forze di dispersione sono deboli ma possono essere importanti quando altri tipi di interazioni sono mancanti o minime (parte (d) della figura \(\PageIndex{5}\)). Questo è il caso della fibroina, la principale proteina della seta, in cui un’alta percentuale di aminoacidi nella proteina ha catene laterali non polari. Il termine interazione idrofobica è spesso usato impropriamente come sinonimo di forze di dispersione. Le interazioni idrofobiche sorgono perché le molecole d’acqua si impegnano nel legame dell’idrogeno con altre molecole d’acqua (o gruppi in proteine in grado di legare l’idrogeno)., Poiché i gruppi non polari non possono impegnarsi nel legame dell’idrogeno, la proteina si piega in modo tale che questi gruppi siano sepolti nella parte interna della struttura proteica, riducendo al minimo il loro contatto con l’acqua.
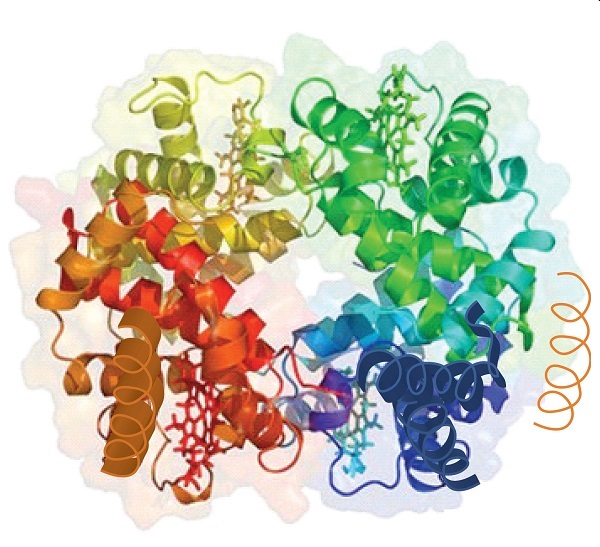
Quando una proteina contiene più di una catena polipeptidica, ogni catena è chiamata subunità. La disposizione di più subunità rappresenta un quarto livello di struttura, la struttura quaternaria di una proteina. L’emoglobina, con quattro catene polipeptidiche o subunità, è l’esempio più frequentemente citato di una proteina con struttura quaternaria (Figura \(\PageIndex{6}\))., La struttura quaternaria di una proteina è prodotta e stabilizzata dagli stessi tipi di interazioni che producono e mantengono la struttura terziaria. Una rappresentazione schematica dei quattro livelli di struttura della proteina è in Figura \(\PageIndex{7}\).
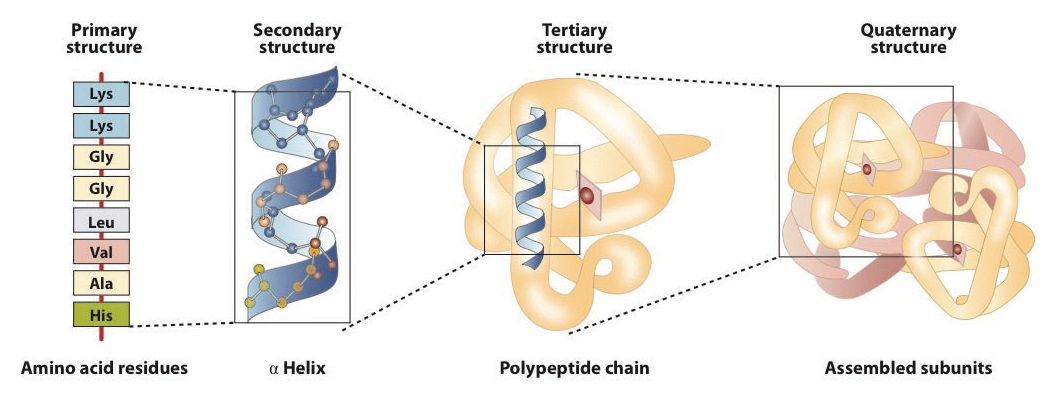
La struttura primaria consiste la specifica sequenza di amminoacidi. La catena peptidica risultante può ruotare in un’α-elica, che è un tipo di struttura secondaria. Questo segmento elicoidale è incorporato nella struttura terziaria della catena polipeptidica piegata., La singola catena polipeptidica è una subunità che costituisce la struttura quaternaria di una proteina, come l’emoglobina che ha quattro catene polipeptidiche.